病毒感染宿主后,其基因组可产生具有功能性的病毒小RNA(small RNA, sRNA),在病毒与宿主相互作用过程中发挥重要作用。然而,目前基于宿主sRNA开发的sRNA识别方法对病毒sRNA的挖掘远远不够,为此,需要一种新的识别方法来提高对sRNA测序数据中病毒sRNA识别的敏感度。
近日,湖南大学生物学院彭友松团队在生物信息学国际权威期刊《Briefings in Bioinformatics》(IF2021=14)在线发表题为“vsRNAfinder: a novel method for identifying high-confidence viral small RNAs from small RNA-Seq data”的研究论文,该研究开发了一种基于sRNA测序数据识别病毒sRNA的生物信息学工具vsRNAfinder,为深入挖掘病毒sRNA提供了帮助。
vsRNAfinder由四个模块组成,包括预处理模块(Preprocessing)、识别模块(Identification)、过滤和注释模块(Filtering and annotation)以及定量模块(Quantification),可在Github上获取(https://github.com/ZenaCai/vsRNAfinder)。该工具通过对病毒参考基因组上的覆盖度进行平滑化处理来降低峰值检测的噪声,进而增强峰值信号,便于峰值检测;同时为了得到高可信度的病毒sRNA,引入了泊松分布模型计算候选sRNA的统计学显著性,进一步提高所识别到的病毒sRNA的可信度。
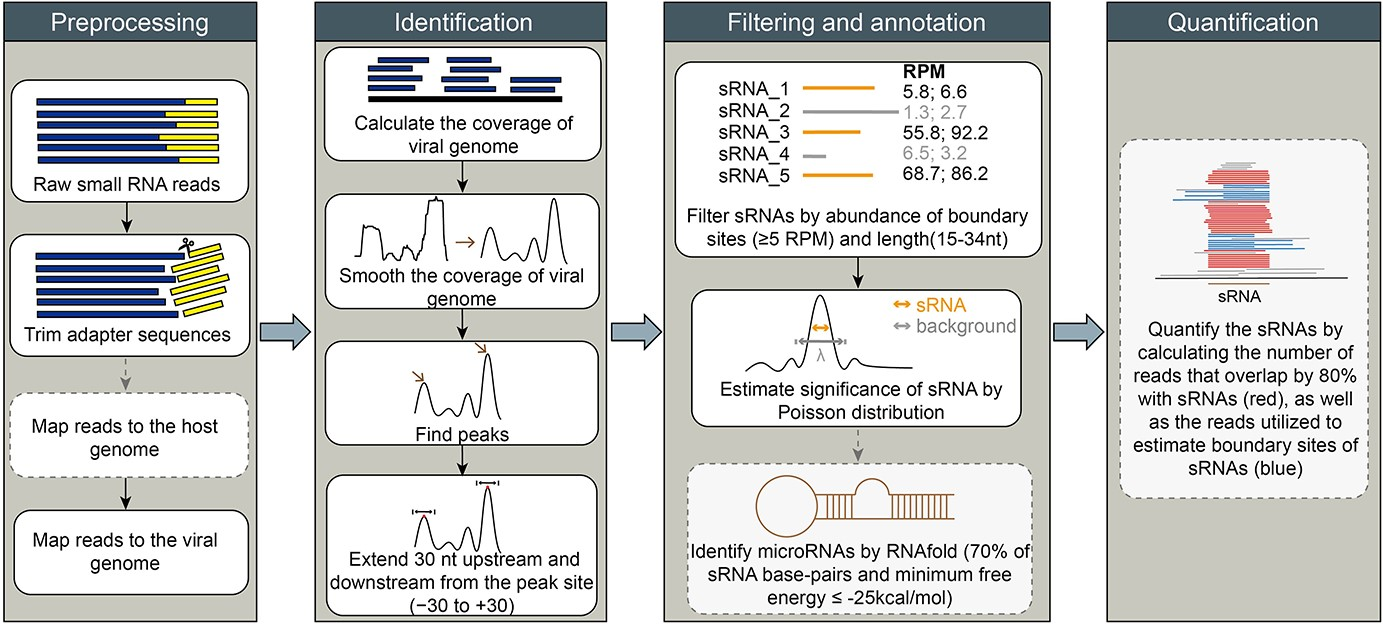
vsRNAfinder的工作流程
该研究进一步将vsRNAfinder与目前广泛使用的sRNA识别工具miRDeep2和ShortStack进行比较,发现vsRNAfinder大大提高了识别病毒sRNA的敏感度(Recall),在综合评分(F1-score)上也优于其他识别工具,同时在运行速度上也快于其他识别工具。由此可见,该研究为病毒sRNA的识别提供了一种有效且快速的方法。
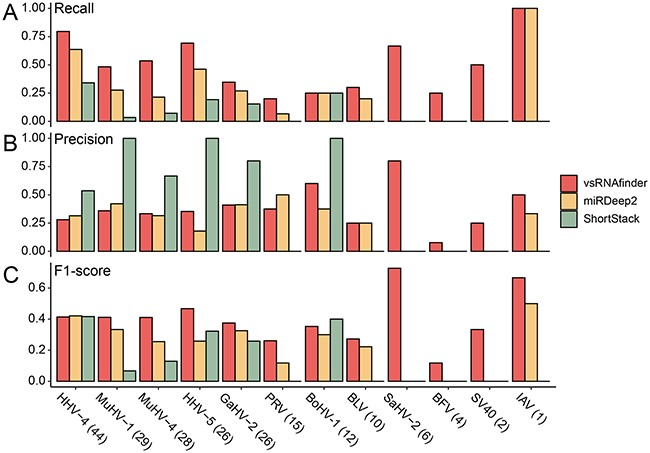
不同工具识别效果对比
该论文的第一作者为湖南大学生物学院的博士研究生蔡泽娜和傅萍,通讯作者为湖南大学生物学院生物信息中心、医学病毒学湖南省重点实验室的副教授彭友松。本研究受到国家自然科学基金(32170651),湖南省自然科学基金(2020JJ3006),湖南大学双一流建设基金(521119400156)等多个项目的支持。
论文链接:
https://academic.oup.com/bib/article-lookup/doi/10.1093/bib/bbac496
论文信息:
Zena Cai, Ping Fu, Ye Qiu, Aiping Wu, Gaihua Zhang, Yirong Wang, Taijiao Jiang, Xing-Yi Ge, Haizhen Zhu, Yousong Peng, vsRNAfinder: a novel method for identifying high-confidence viral small RNAs from small RNA-Seq data, Briefings in Bioinformatics, bbac496, https://doi.org/10.1093/bib/bbac496
